|
天然产物是来源于微生物、植物和动物的一类结构高度多样的化学物质,在新药发现、化学生态和代谢组学等领域研究中占据重要地位。尽管实验室自动化以及分离鉴定技术的快速发展显著提升了天然产物的发现效率,但其结构解析仍然高度依赖人工经验和多维谱学验证,过程复杂、周期漫长,已逐渐成为制约天然产物研究与应用开发的关键瓶颈。
中国科学院昆明植物研究所植物化学与天然药物全国重点是实验室邱明华研究团队近年来持续聚焦于基于深度学习方法的天然产物结构与活性研究。在前期工作中,团队系统梳理并深入讨论了机器学习辅助光谱解析在天然产物研究中的一系列前沿技术(Natural Product Reports, 2023, 40, 1735-1753),为该领域的进一步发展奠定了有价值的理论框架和方法基础。
核磁共振(NMR)是天然产物结构鉴定中最重要的分析工具之一。然而,NMR 谱图的结构注释通常高度依赖成本高昂的实验数据。尽管近年来基于量子化学计算或者人工智能算法生成的虚拟NMR数据在一定程度上扩展了数据库规模,但基于虚拟数据库的结构注释准确率仍然有限。为解决这一问题,研究团队近期提出了一种新的工具 VirMolAnalyte(https://www.virmolanalyte.top/),可在无实验数据依赖下,实现对 ¹³C DEPT NMR 谱图的高质量结构注释。
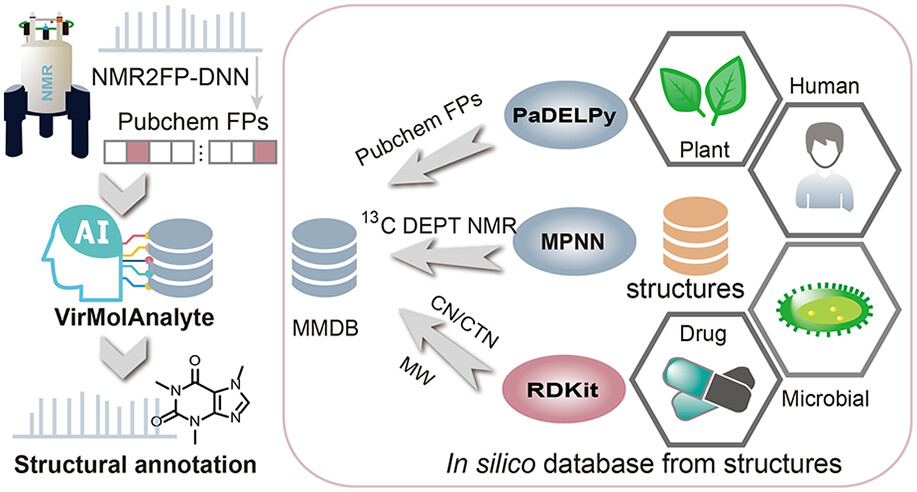 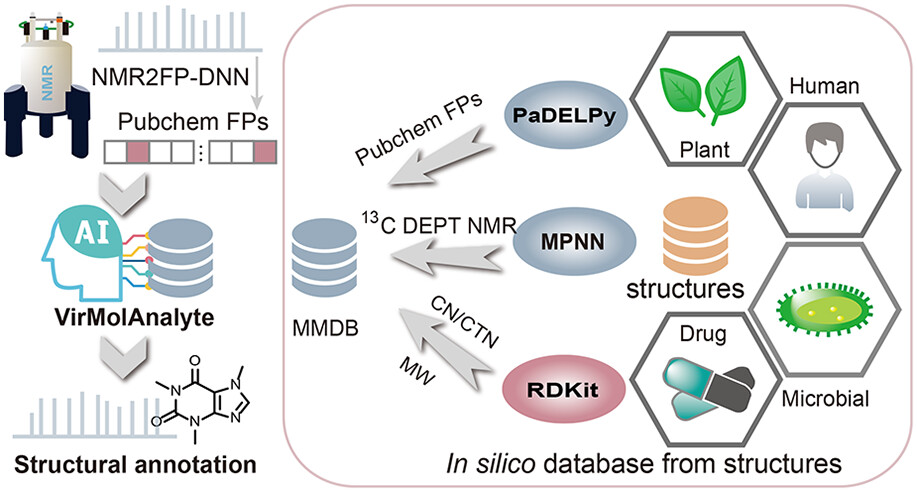
图1 基于 ¹³C DEPT NMR的注释天然产物化学结构新工具VirMolAnalyte的思路
VirMolAnalyte 利用深度神经网络从 ¹³C DEPT NMR 谱中提取分子指纹信息,并结合“筛选–评估”(filter–evaluator)策略开展多维 in silico 数据库检索。其核心思想在于改进了基于化学位移匹配的搜索算法,同时将基于深度神经网络预测的分子指纹信息融合到化学位移检索过程中,从而显著提升基于虚拟数据库的准确率。在DB6123数据集上的评估结果表明,该融合策略的Top 1准确率达到 94.2%,明显优于传统的化学位移搜索方法。
在此基础上,研究团队整合了 COCONUT、CMAUP v1.0、PMhub、HMDB、NPAtlas 和 FooDB 等多个涵盖植物、人体、微生物的代谢产物及药物来源的结构数据库,构建了一个包含约 60 万个天然产物结构的综合多维信息数据库MMDB。在基于MMDB的结构注释任务中,VirMolAnalyte 仍取得了超过90.0%的Top 1结构注释准确率,表明该融合策略在大规模复杂数据库场景下具有良好的泛化能力。另外,为验证 VirMolAnalyte 在新天然产物结构解析中的应用潜力,研究团队将其用于分析来源于阿拉比卡咖啡(Coffea arabica L.)果皮的一种未知代谢物。检索结果未匹配到已知化合物,但成功识别出与咖啡二萜类相关的关键结构单元,提示其包含三环醚和五元内酯等结构特征。结合二维 NMR 数据及后续构型分析,最终确认该化合物为一种罕见的螺环型咖啡二萜类新化合物,初步表明 VirMolAnalyte 能够为新颖天然产物的初期结构推测和确证提供有效线索。
VirMolAnalyte 工具弥补了NMR数据在化合物初期结构鉴定阶段缺乏高质量虚拟数据库注释方法的空白,为后续精细结构解析提供方向指引,有助于提升天然产物研究和新药发现的前期效率。相关研究工作在线发表于Analytical Chemistry (2025, 97, 51, 28181-28191)。中国科学院昆明植物所邱明华研究员为通讯作者,胡贵林博士后和Jameel Hizam Alafifi博士为论文共同第一作者,研究工作获云南省科技重点专项(202003AD150006)、重大专项(202305AH340005),中国科学院B类先导专项(XDB1230201),国家自然科学基金青年基金项目(82504632),国家博士后研究人员计划(GZC20232766)等项目资助。
文章链接
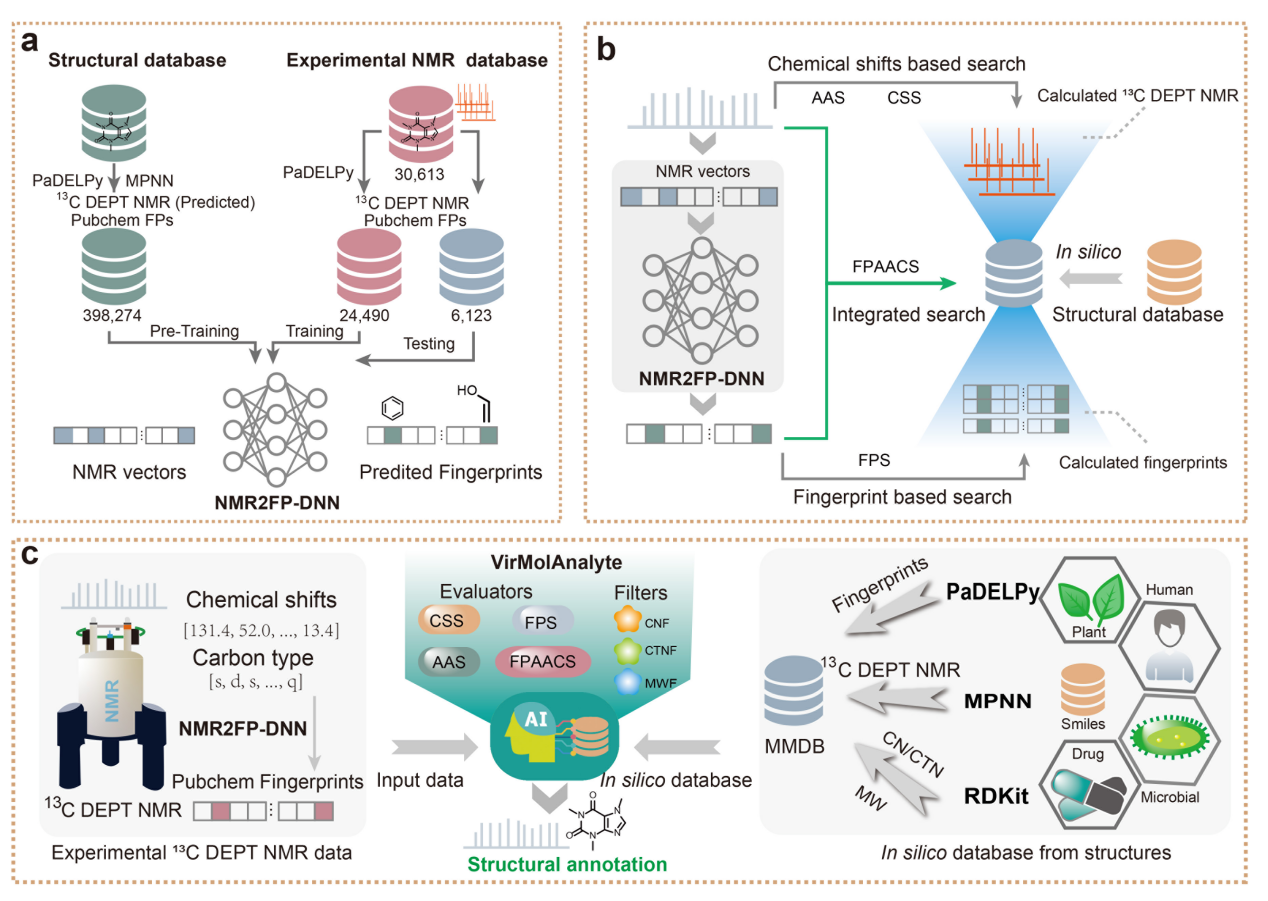 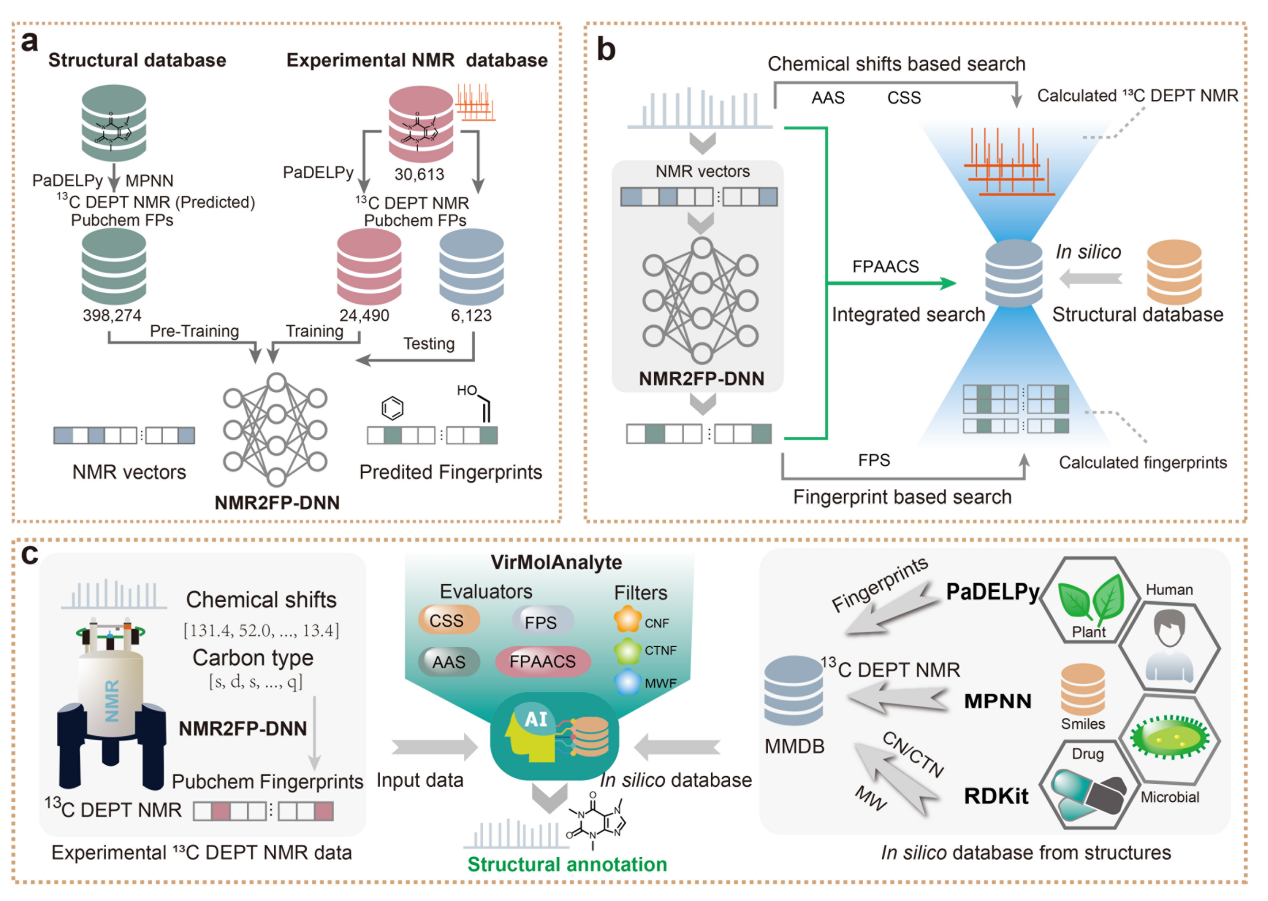
图2 基于“筛选-评估”策略的 VirMolAnalyte。(a) NMR2FP-DNN 模型的训练流程,用于从13C DEPT NMR 谱中预测分子指纹;(b) VirMolAnalyte 中各评估器的基本原理;(c) VirMolAnalyte 的整体工作流程:NMR2FP-DNN 根据输入的13C DEPT NMR 数据预测 PubChem 分子指纹,预测指纹与化学位移信息共同作为输入,通过“筛选–评估”策略在 in silico 数据库中完成结构注释。
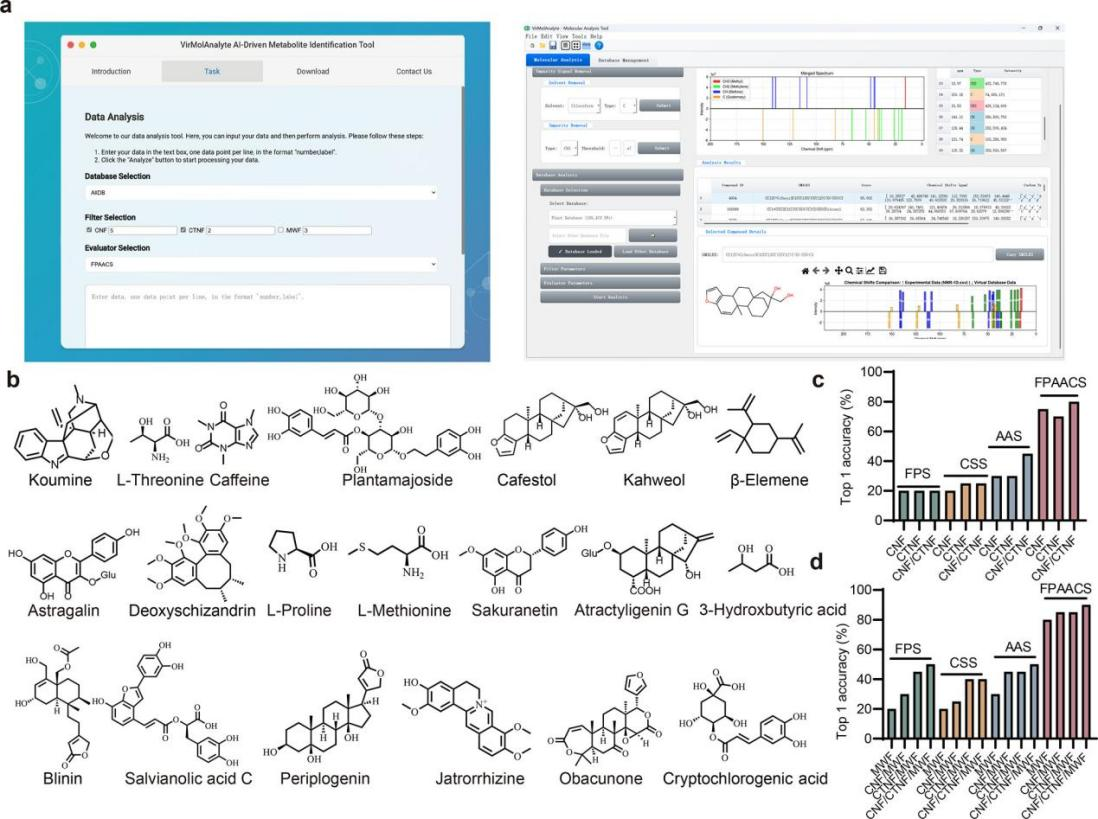 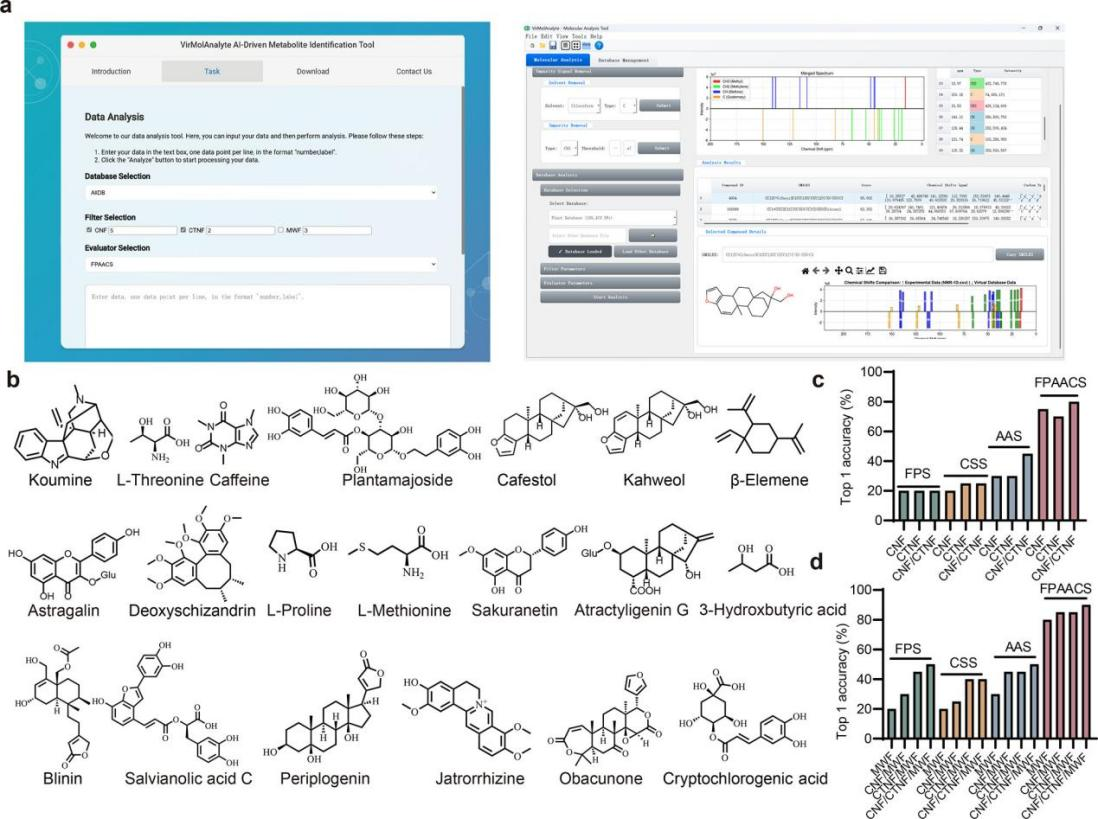
图3 VirMolAnalyte 工具基于 MMDB 数据库的天然产物结构注释
|

